
История
Часть товаров из корзины будет перемещена в статус отложенных и не сможет быть оформлена для заказа, если вы продолжите работу в данном регионе
История

История
История
 Развитие технологии массового параллельного секвенирования NGS в последнее 10-летие значительным образом расширило возможности исследований в фундаментальной биологии и медицине, прежде всего, за счет снижения стоимости секвенирования на несколько порядков по сравнению с секвенированием по Сэнгеру.
Развитие технологии массового параллельного секвенирования NGS в последнее 10-летие значительным образом расширило возможности исследований в фундаментальной биологии и медицине, прежде всего, за счет снижения стоимости секвенирования на несколько порядков по сравнению с секвенированием по Сэнгеру.
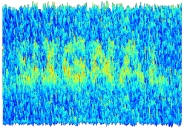
Несмотря на значительное улучшение производительности приборов, уровень ошибок NGS секвенирования практически не изменился. Точность всех аналитических измерений, включая секвенирование ДНК, зависит от соотношения между истинным значением и значением, полученным с помощью метода обнаружения. Это аналогично съёмке на цифровую камеру: при низком соотношении сигнал/шум, изображение нечеткое (Рис.1), но с повышением качества сенсора (Рис.2) появляется возможность увидеть сигнал от истинных объектов и снизить неспецифический шум. Для приложений NGS, требующих высокой точности, были разработаны методы фильтрации данных, способы уменьшения ошибок в консенсусной последовательности, однако фоновые ошибки самих платформ секвенирования второго поколения по-прежнему ограничивают обнаружение низкочастотных вариантов.
 |
 |
 |

|
| Рис. 1 |
Рис. 2 |
В настоящее время превалирующий подход секвенирования в NGS — это секвенирование путем синтеза SBS (sequencing by synthesis), в котором прочтение матричной цепи происходит вследствие детекции определенного сигнала от встраивания нуклеотидов во время синтеза цепи. За исключением технологии PacBio, где синтез цепи мониторируется постоянно, во всех остальных SBS подходах процесс синтеза построен на циклических реакциях.
Все эти подходы могут быть далее разделены на 2 основные категории в зависимости от используемой флоуграммы: циклическая обратимая терминация CRT (Cyclic reversible termination. Напр.: Illumina/Solexa, Direct/Helicos) и присоединение единичных нуклеотидов SNA (Single Nucleotide Addition; Напр.: Roche/454, Ion Torrent).
В CRT подходах 3′-OH заблокирована и, следовательно, если нуклеотид был встроен в комплементарную цепь, дальнейшего удлинения цепи не происходит до отделения метки. В каждом реакционном цикле подается 4 нуклеотида, ковалентно сшитых со своими флуоресцентными метками, и только одна база, удлинившая цепь, может быть определена в цикле.
В подходе SNA в каждом цикле в реакцию вводится только один нуклеотид с открытой 3′-OH группой. Элонгация происходит, когда предлагаемый нуклеотид комплементарен матрице.
У обоих подходов есть свои преимущества и недостатки. CRT используют флуоресценцию, которая стабильна, высокоэффективна в плане сигнализирования о встроившемся нуклеотиде. Однако комплексность и несовершенство реакционной химии, вызывающее молекулярное шрамирование (molecular scars) на растущей цепи после удаления флуоресцентной метки, приводит к ошибкам в секвенировании и лимитирует длину прочтения.
Большим преимуществом SNA является то, что продуктом реакции является нативная ДНК без терминирующих групп или шрамов, оставленных отщепившимися метками, в теории давая возможность получения более длинных ридов. Однако детектируемый сигнал в современных приборах по технологии SNA транзиентный и имеют плохую линейность между интенсивностью сигнала и числом dNTP, включенных в один цикл реакции в гомополимерных областях.
|
Cygnus Biosciences
Система для высокопроизводительного секвенирования S100 |
||||||||||||
|
111-001-101
|
|
|
New
|
|
По запросу По запросу |
|
|
|
||||
|
S100 — это настольный секвенатор, отличающийся компактным размером, интуитивно понятным управлением, высокой точностью секвенирования, гибкостью адаптации к различным сценариям и широким спектром выполняемых задач. Единственный прибор для массового параллельного секвенирования, в котором внедрен алгоритм коррекции ошибок (Error Correction Code), что позволяет повысить точность секвенирования в 10 раз. Генерации кластеров ДНК и секвенирование осуществляются в одном приборе, без выполнения ручных операций. Два режима секвенирования: сверхбыстрый (BitSeq), подходящий для определения количественной структуры цепи (делеции, дупликации, небольшие CNV) и сверхточный (ECC), подходящий и для детекции низкочастотных вариантов. Для удобства управления системой имеется встроенный сенсорный экран. Секвенатор также имеет полный набор программного обеспечения (с возможностью обновления) для управления системой и первичной обработки данных и встроенный аппаратный модуль для ускорения вычислений. Основные применения секвенатора S100
Характеристики секвенатора S100
Комплектация:
|
||||||||||||
|
|
||||||||||||
Команда ученых из Университета Пекина решила кардинально поменять используемую традиционно реакционную химию и взять лучшее от обоих подходов прочтения последовательности, предложив новую стратегию секвенирования с уникальным алгоритмом коррекции ошибок (ECC error-correction code). Два типа нуклеотидов подаются в каждом цикле, у синтезируемой цепи открыта свободная 3′-OH группа, которая удлиняется до тех пор, пока ни одного нативного нуклеотида в смеси не может быть встроено. Несмотря на то, что каждая такая реакция обеспечивает получение информации о строении вырожденной цепи с частично определяемым составом оснований, три последовательных ортогональных прочтения матричной цепи дадут информацию о ее строении с беспрецедентной точностью. Алгоритм коррекции ошибок ECC элиминирует большинство ошибок секвенирования, а в сочетании с запатентованной высокоэффективной флуорогенной химией позволяет исключить все ошибки в последовательности длиной 200 п.о.¹
Высокоточный метод секвенирования особенно необходим для обнаружения мутаций в фетальной ДНК в материнской крови, идентификации редких вариантов в циркулирующей опухолевой ДНК или в высокогетерогенных опухолевых тканях. Точность, характерная для доступных в настоящее время NGS платформ, не достаточна для этих приложений, поэтому на практике прибегают к сложным стратегиям подготовки образцов и соответствующему биоинформатическому анализу. Технология ECC дает возможность существенно улучшить качество данных и соответствовать требованиям точности прецизионной медицины.
Основные публикации, описывающие технологию:
1. Zitian Chen et all. Highly accurate fluorogenic DNA sequencing with information theory–based error correction. Nature Biotechnology. 2017.
2. Zitian Chen et all. Fluorogenic Sequencing Using Halogen-Fluorescein Labeled Nucleotides. ChemBioChem. 2015.
3. Wenxiong Zhou et all. A Fuzzy sequencer for rapid DNA fragment counting and genotyping. bioRxiv. 2023. doi.org/10.1101/2023.10.24.563729.
4. Wenxiong Zhou et all. A virtual sequencer reveals the dephasing patterns in error-correction code DNA sequencing. National Science Review. 2021. doi: 10.1093/nsr/nwaa227
См. также:
Станции для автоматической подготовки библиотек ngsС помощью личного кабинета Вы сможете:
Сравнение